EpiTYPER DNA 甲基化分析原理
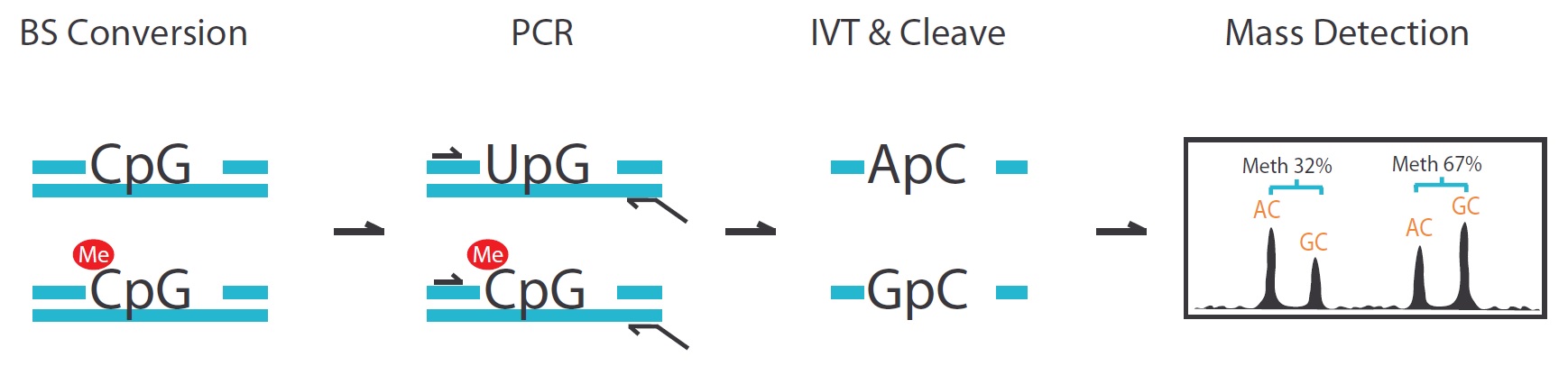
包含四個反應步驟:Bisulfite conversion、PCR、MassCLEAVE (T7-IVT and Base-specific RNA cleavage)及Mass detection。
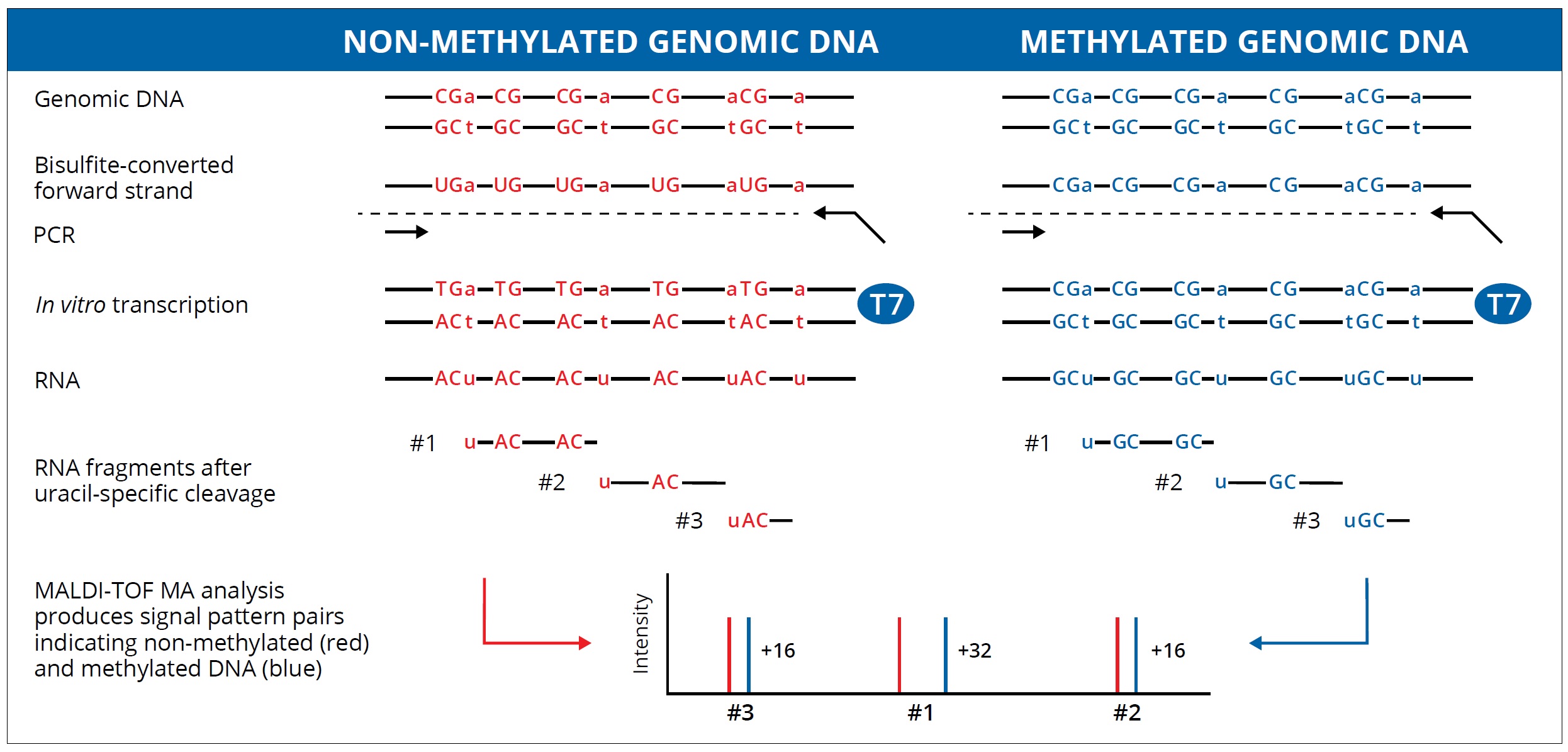
- Bisulfite conversion 後,未甲基化之 C 將被轉換成 U,而甲基化的C鹼基將維持 C 之型態。
- 經 PCR 反應增幅時,所使用的 reverse primer 因帶有 T7 RNA polymerase promoter 序列,此時 T7 RNA polymerase 可利用此序列進行 in vitro transcription 合成出 RNA,並同時將序列中的 CTP 置換成為 dCTP。
- RNA 產物再由 RNase A 進行 Base-specific cleavage。RNase A 能截切 RNA 序列中的 C 與 U 的位置,由於 CTP 已事先置換成 dCTP,因此RNase A 將只會專一性的截切 U 之位置。
- 整個序列通常會被截切成數十個片段,片段中如果帶有 CpG site,會因為甲基化狀態出現出兩種序列 ( GC 或 AC ),此兩種序列分別表示甲基化或者是未甲基化的DNA。在質譜分析可以觀察到兩種序列為分子量大小相差 16Da 的訊號,根據訊號波峰面積比率即可推算出該 CpG site 甲基化比例。
特色與優點:
- 質譜偵測技術提供精準的甲基化定量結果
- 分析數據CV< 5%,定量線性範圍5%~95%
- 單一反應最長分析達600bp之區域
- 不需定序、可快速分析
- 優異的花費效益與樣本容載率
分析服務流程與方式:
- 客戶準備序列資訊
- 本公司進行Primer設計與實驗評估,並寄送設計結果報告
- 客戶根據設計的primer序列進行PCR測試
- 客戶提供成功的膠圖結果給本公司評估是否可進行實驗
- 客戶準備Bisulfite PCR產物送件
- 本公司進行後續實驗分析
- 產出結果報告
分析服務檢體需求:
接受檢體形式 : Bisulfite PCR產物
最少樣本數量 : 24 reactions
樣本總需求量: 10μl Bisulfite PCR產物
須注意:
- 膠圖上的樣本必須為 single band
- 若膠圖顯示 primer dimer 嚴重,會影響實驗成功機率
- 請使用豐技實驗室建議的 primer進行PCR